RNA-seq连特异性
The strandness of RNA-seq analysis
前段时间一直在研究关于illumina TrueSeq stranded RNA-seq中的strand如何判断的问题。之后我又查了很多资料,终于弄懂了。现在写下来,如果我有错误,欢迎继续指正。 以下文字和图片的引用链接都已经给出,如果图片在邮件中无法显示,可以打开链接的。
先说结论:对于Illumina TrueSeq stranded RNA protocol,主要采用的是dUTP method。RNA的strand是与read1(先测的read)相反,与read2(后测的read)的strand相同的。换句话说,如果read1比对到基因组正链上,则对应的RNA是在基因组的负链上。如果read2比对到基因组正链上,则对应的RNA应该是比对到正链上。
具体解释如下:
1.关于strand:
1)DNA是由两条互补配对的链组成的。按照预定俗称,把其中一条链称为正链(plus strand or forward strand),另一条则是负链(minus strand or reverse strand),这个定义完全是人为的。我们下载的hg18、hg19、hg38的fasta格式只给出了正链的序列。另一条链可以根据互补配对得出。
2)我们读取的链的时候,经常是从5’到3’的,无论DNA还是RNA。测序出来的两个read,也都是从5’到3’的。视觉上,我们读取plus strand的时候,from left to right。读取minus strand的时候,from right to left。
3)RNA会根据一条DNA模板合成,我们在IGV或者UCSC上会看到RNA的链的信息,如果一条RNA在正链上(如下图),其实该条RNA是以负链作为模板形成的,但是该条RNA与正链的序列一直。我们把和合成RNA时的DNA模板的链称为template strand或者antisense strand。把与RNA序列相同的DNA的链称为coding strand或者sense strand。sense/antisense, coding/template strand可以在基因组的plus strand也可以在基因组的minus strand。
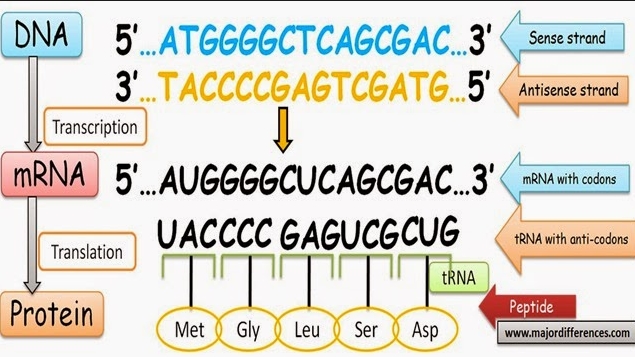
http://www.majordifferences.com/2015/01/difference-between-sense-and-antisense.html
2、illumina测序之sample preparation:
1)illumina测序的sample preparation的步骤,一般是讲DNA或者是cDNA打断,然后repair ends,add A at 3’end (为了和Y-shaped adaptor结合),然后加上adaptor。
2)adaptor也是由两条链组成的(universal adaptor和indexed adaptor)。unversal adaptor的3’端有一个T,为了可以和DNA fragment上的A互补。universal adaptor的3’端和indexed adaptor的5’端有12bp的碱基互补配对,使得他们两个可以呈现Y字形。indexed adaptor的中间有6个bp的indexed sequence,可以用来在同一个land中有多个样本时做区分。
3)若是非连特异性的,对于最后合成的sample,中间的DNA fragment既可以来自正链,也可以来自负链。
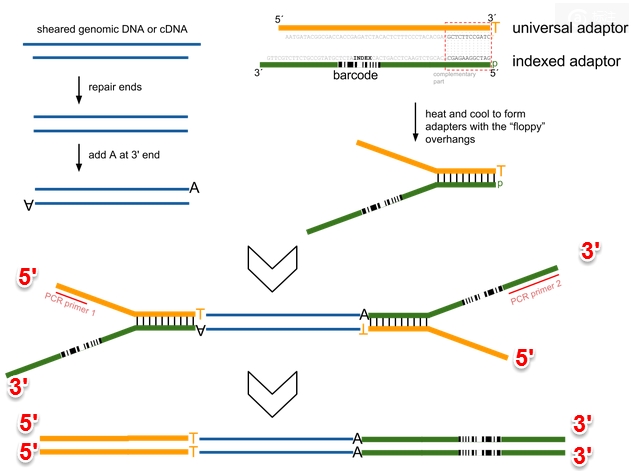
http://onetipperday.blogspot.com/2013/06/illumina-hiseq2000-adaptor.html
3、dUTP method的sample preparation:
dUTP metod主要是在反转完first-strand cDNA之后,在合成second-strand cDNA的时候,用dUTP代替了dTTP,之后加完adaptor,用UDGase处理,将second-strand cDNA消化掉。这样最后合成的sample,中间的DNA fragment一定是first-strand cDNA。这也是为什么对于dUTP数据处理,tophat的参数应该为“–library-type fr-firststrand”。注意,这里的first-strand cDNA与RNA strand(或者基因组上的coding strand)相反。
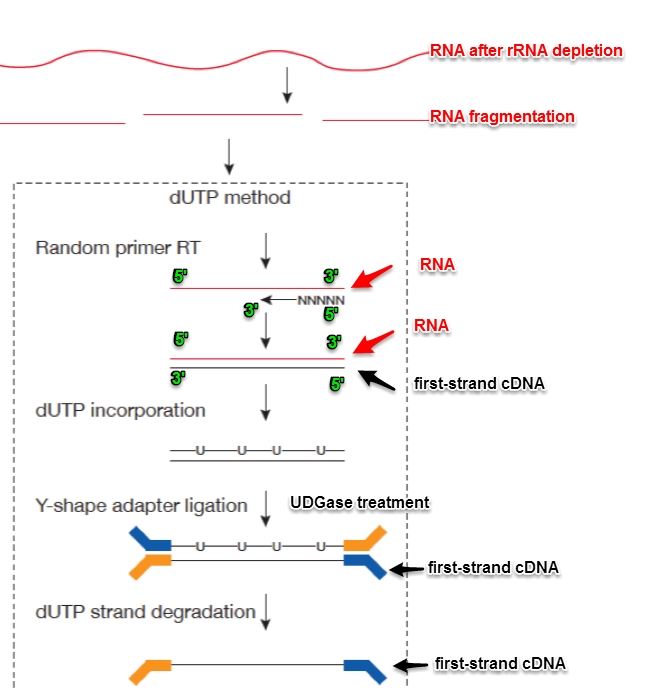
http://onetipperday.blogspot.com/2013/06/illumina-hiseq2000-adaptor.html
4、illumina测序之cluster generation和sequencing
1)sample preparation之后,样品就上机测序了。在flow cell的表面,固定着很多个DNA序列,他们有两种P5和P7,均是5’在flow cell的表面,3’露在上面。
2)P5的序列与sample 5’端的unversial adapter 开始的44(数字可以不同)个bases相同,P7的是与sample的3’端的indexed adapter的24(数字可以不同)个bases互补配对。P5和P7同时可以充当与sample DNA序列杂交的探针还有PCR扩增的引物。
3)sample DNA上机后,首先与P7杂交,然后以P7为引物合成。如下图,之后洗掉原来的sample DNA,然后P7上的DNA的3’端正好与P5杂交,建起bright,然后再以P5为引物合成。就这样不断amplification。
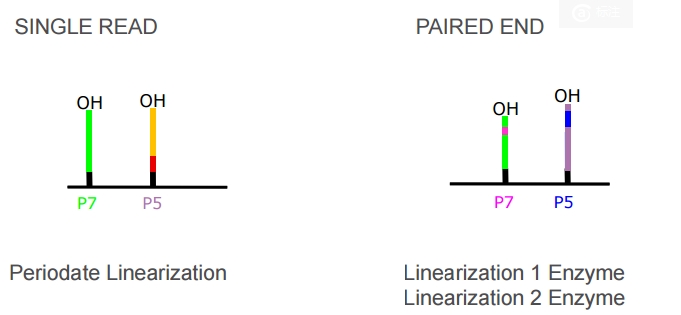
4)之后测序的时候,会先将P5进行Periodate linearization(具体不是很懂,应该就是把P5上的DNA弄走)。然后用引物来测定P7中间DNA fragment的序列,就是read1中的序列(注意,这里的引物与P5和P7不同,可以保证直接测出中间感兴趣的序列。但是若中间DNA fragement太短,就会测到adaptor序列,这就是为什么fastq文件要做adaptor trimming)。如果是Pair-end序列。P7上的序列测完之后,再合成P5的DNA,然后再用另一种酶把P7弄走,去测P5的序列。这样就保证测量了pair-end。从这个过程可以看到read1和read2一定是分布会比对到基因组不同的链上。
5)对于strand-specifc,由于sample DNA中间感兴趣的DNA fragement只有first-strand。这个fragment先于P7杂交,然后合成second strand cDNA。然后测序的时候,read1是与P7上的second strand cDNA互补配对的。(first-strand cDNA <- rc -> P7上的second strand cDNA <-rc->read1的序列)。故read1与first-strand cDNA的链相同,与RNA的strand链相反。反之,read2与RNA strand的链相同。
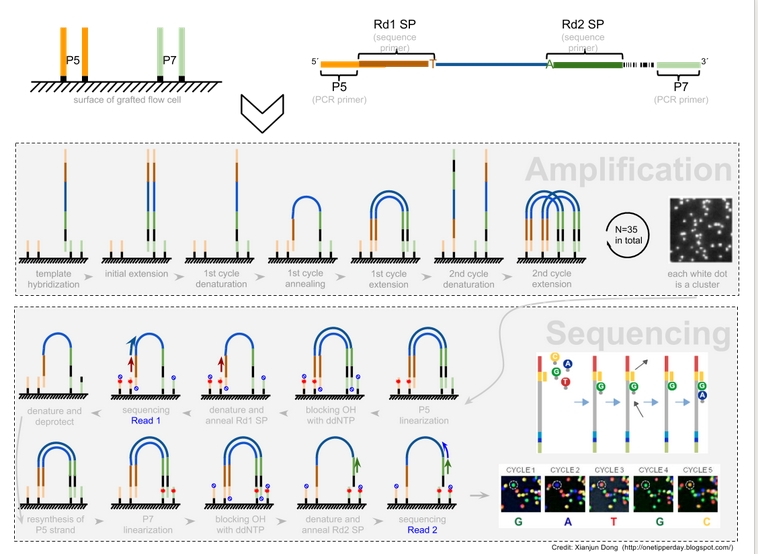
http://onetipperday.blogspot.com/2013/12/illumina-hiseq2000-adaptor-and.html
http://skatebase.org/sites/skatebase.org/files/workshops/W1_IntroIlluminaNGS.pdf
5、tophat中output的bam文件中有一个tag XS是错的
tophat输出的bam文件的attribute tag中有一个XS是指示RNA所在的strand的。但是很多人都发现了错误。而且Encode还写了一个perl脚本来修改XS tag。也有人说XS主要是根据splite site的序列判断的。XS: “+” for GT-AG and “-“ for CT-AC。另外,因为cufflinks是需要XS的这个tag的,所以这个问题还需要再研究一下。
6、可以通过sam文件的第2列的flag来判断read的strand
sam flag可以指示该条read是read1(first in pair)还是read2(second in pair),或者比对到正链上(mate reverse strand)还是负链上(read reverse strand)。 我其实也不懂得flag这个16进制,这有个网址给了一个简单的软件,可以帮助计算。然后用samtools view -f或者-F就可以根据flag来筛选了。
7、参考资料:
http://onetipperday.blogspot.com/2013/06/illumina-hiseq2000-adaptor.html
http://onetipperday.blogspot.com/2012/07/how-to-tell-which-library-type-to-use.html
http://onetipperday.blogspot.com/2013/12/illumina-hiseq2000-adaptor-and.html
http://www.personal.psu.edu/iua1/courses/illumina-sequencing.html
http://skatebase.org/sites/skatebase.org/files/workshops/W1_IntroIlluminaNGS.pdf